
特聘研究员
zbliang@szbl.ac.cn
2021至今 深圳湾实验室 特聘研究员
2017-2021 美国耶鲁大学 博士后
2015-2017 美国耶鲁大学医学院 博士后
2009-2015 美国密西根大学 分子生物学博士
2004-2008 上海交通大学 植物生物技术学士
研究领域
课题组主要从事基因组不稳定性机理研究及新型基因组编辑工程技术开发,同时运用前沿合成生物学手段以阐明人体疾病、衰老机理、发现新颖天然药物及靶点、以及研发基于基因、免疫和干细胞治疗等精准医学疗法。课题组具有很强的学科交叉背景,开展课题广泛涉及分子细胞生物学、遗传学、生物化学、系统生物学、合成生物学、生物信息、生物技术、血液免疫学、生物医学工程、病毒学等学科。实验室能灵活运用多种生物学模型,涵盖病原微生物、病毒、植物、酵母、哺乳动物细胞系、小动物模型以及体外无细胞体系等。实验室将开展从基础医学研究到精准医学技术开发,再到临床转化应用的一系列工作。
主要研究方向:
(1)新一代基因组编辑工程技术开发与精准医学应用
(2)基因组不稳定性在癌症及衰老等疾病中的致病分子机理
(3)高效天然药物发现及生物合成创新技术平台
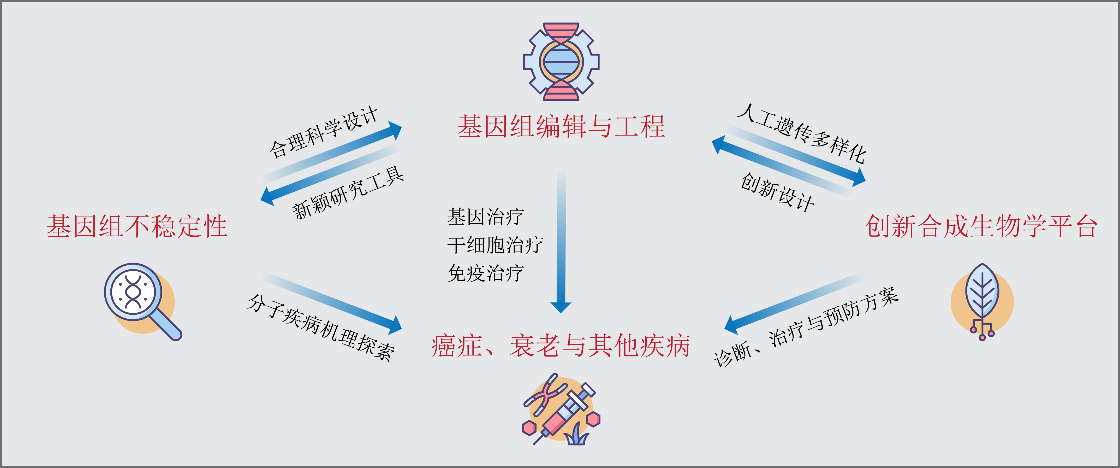
注解:梁卓斌课题组将开展多研究领域的交叉融合以促进精准生物医药基础研究与临床转化应用
成果
课题组PI在旅美期间先后以PI或Co-PI身份主持参与了总值约100万美金的多个研究项目,发表高水平论文十余篇。以第一作者或共同通讯作者论文发表在Nature Communications, Nucleic Acid Research, Cell Report, Plant Physiology 等杂志;以共同作者身份在Nature Biotechnology, ACS Central Science, Nature Communicates, PLOS Genetics, Journal of Biochemistry 等杂志发表多篇论文。主要成果如下:
1. 揭示了转基因植物遗传转化过程中宿主DNA修复途径参与农杆菌单链T-DNA双链化过程的新机理。
2. 成功开发出可用于多基因天然药物生物合成的新型技术平台,并实现世界上首次抗疟疾药物青蒿素在转基因烟草中的全生物合成。
3. 阐明基因编辑工具Zinc Finger Nuclease(ZFN)产生的5’端突出DNA双链断裂(DSB)末端结构影响突变产生的分子遗传学机理,对类似基因编辑工具开发应用及阐明DSB非同源末端连接(NHEJ)修复缺陷的致病机理产生积极意义。
4. 发现范可尼贫血症(Fanconi Anemia)相关的DNA链间交联(ICL)修复蛋白复合体FANCI-FANCD2可通过结合多种RNA底物产生特异位点泛素化修饰,揭示了其参与修复RNA转录引起的基因组不稳定性在相关血液疾病的新机理。
5. 大幅改良新型真核细胞基因组编辑工具eMAGE的编辑效率和编辑精度,并应用于人类乳癌等癌症相关受体蛋白致病突变的研究。
6. 开发出一系列可用于快速构建并安全检测新型冠状病毒(SARS-CoV-2)泛基因组突变的基因组工具,有望被广泛应用于新冠COVID-19致病机理研究、治疗药物筛选以及针对病毒变异的疫苗研发。
荣誉
• 2003 国家一级运动员(国际象棋)
• 2005 上海交通大学最高A等奖学金
• 2007 上海交大优秀学生干部(曾任学院学生会主席、学生科创中心主任)
• 2008 上海市奖学金
• 2008 上海市优秀毕业生
• 2015 美国密西根大学校长Coleman奖学金
• 2015 美国密西根大学博士论文奖金
• 2016 Damon Runyon博士后奖金
• 2018 耶鲁癌症中心创新奖
• 2020 耶鲁大学新冠病毒先导研究奖励
媒体报道
2. Damon Runyon Cancer Research Foundation
3. Awarded NSF-EDGE funding to develop cutting-edge genomics tools – as Co-PI in the Isaacs laboratory
4. Yale COVID-19 Pilot Projects – as Co-PI in the Isaacs laboratory
代表论文
1. Liang Z.*, Metzner E. and Isaacs F.J.* (2020). “Advanced eMAGE for highly efficient combinatorial editing of a stable genome.” bioRxiv doi.org/10.1101/2020.08.30.256743 (Nat Commun, in revision) (IF=12.1)
2. Liang Z., Liang F., Teng Y., Chen X., Liu J., Longerich S., Rao T., Green A. M., Collins N. B., Xiong Y., Lan L., Sung P. and Kupfer G. M. (2019). “Binding of FANCI-FANCD2 complex to RNA and R-Loops stimulates robust FANCD2 monoubiquitination.” Cell Rep (IF=8.1)
3. Liang Z., Sunder S., Nallasivam S. and Wilson T. E. (2016). “Overhang polarity of chromosomal double-strand breaks impacts kinetics and fidelity of yeast non-homologous end joining.” Nucleic Acids Res 44(6): 2769-2781. (IF=11.5)
4. Liang Z. and Tzfira T. (2013). “In vivo formation of double-stranded T-DNA molecules by T-strand priming.” Nat Commun 4: 2253. (IF=12.1)
5. Liang Z.*, Chiruvella K. K.*, and Wilson T. E. (2013). “Repair of double-strand breaks by end joining.” Cold Spring Harb Perspect Biol 5(5): a012757 – Chapter XVI of the Book: DNA Repair, Mutagenesis, and Other Responses to DNA Damage. (*co-first author)
个人主页:https://scholar.google.com/citations?user=DVIPilkAAAAJ&hl=en