
特聘研究员
longhaizhen@szbl.ac.cn
2022 至今 深圳湾实验室 特聘研究员
2020-2022 中国科学院生物物理研究所 副研究员
2018-2020 中国科学院生物物理研究所 助理研究员
2016-2018 中国科学院生物物理研究所 博士后
2010-2016 中国科学院大学生物物理研究所 博士
2006-2010 湖南师范大学 学士
研究领域
DNA复制是遗传信息精确传递的基础,受到遗传因素和表观遗传因素的共同调控。在基因组中,DNA复制是从某些特殊的区域开始的,该区域被称为“复制起始位点”。正确的DNA复制起始是生理状态下细胞进行生长分裂和发挥生理功能的基础,但是,在位置或时间上发生错误的DNA复制起始都可能会导致DNA序列的突变以及基因组不稳定性,甚至遗传信息的错误传递,从而促进癌症的产生以及遗传疾病的发生。
基于此,本课题组的研究方向是DNA复制的表观遗传调控机制及其在肿瘤发生发展和细胞分化发育过程的作用。
成果
龙海珍研究员主要围绕表观遗传因子在复制和转录过程中的生物学功能进行了一系列研究,研究成果发表在Nature、Nature Structural & Molecular Biology、BMC Biology以及Genes & Development、Genome Biology 等杂志上。研究成果荣获2020年度中国科学十大进展(进展四:揭示人类遗传物质传递的关键步骤),并入选科技部“十三五”代表性成果展。还获得了深圳市优青、中国科协青年人才托举工程,科技部国家重点研发计划,国自然青年基金,中科院青年创新促进会等项目的支持。
主要成果如下:
(1)多细胞真核生物中,DNA复制起始位点的识别机制仍不清楚,课题组PI的研究工作揭示了一个新颖的由组蛋白变体H2A.Z介导的DNA复制表观遗传调控通路——H2A.Z-SUV420H1-H4K20me2-ORC1,相关成果发表在Nature杂志,获F1000 prime推荐,并荣获2020年度中国科学十大进展(进展四:解释人类遗传物质传递的关键步骤),以及国家“十三五”科技创新成就展。同时课题组PI与结构生物学、化学等方向专家合作,进一步探究了该通路的转化应用前景。
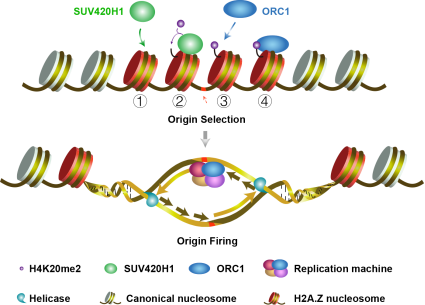
组蛋白变体H2A.Z在DNA复制起始位点选择和激活中的作用
(2)研究发现组蛋白 H4S47 的O-GlcNAc 修饰能通过促进蛋白激酶 DDK 在染色质上的招募,帮助解旋酶 MCM 复合物完成激活,进而开启 DNA 的复制。该研究将DNA复制与细胞代谢也联系起来,为深刻理解细胞如何在动态变化的营养环境下保障遗传物质的正确传递开启了新的视角。相关研究成果发表在Nature Structural & Molecular Biology杂志。
(3)复制、转录、翻译是中心法则的核心内容。课题组PI研究了表观遗传在基因转录调控中的作用,并发现组蛋白变体H2A.Z和H3.3能够协同调控染色质高级结构的动态变化,从而调控基因转录,并进一步参与了免疫等重要生理过程。
荣誉
• 2023 深圳市优秀科技创新人才培养项目(优青项目)
• 2022 入选中国科协生命科学学会联合体第七届“青年人才托举工程”项目
• 2020 中国科学院青年创新促进会会员
媒体报道
代表论文:
(1) Haizhen Long#; Liwei Zhang#; Mengjie Lv#; Zengqi wen#; Wenhao Zhang; Xiulan Chen; Peitao Zhang; Tongqing Li; Luyuan Chang; Caiwei Jin; Guozhao Wu; Xi Wang; Fuquan Yang; Jianfeng Pei; Ping Chen; Raphael Margueron; Haiteng Deng; Mingzhao Zhu*; Guohong Li*; H2A.Z facilitates licensing and activation of early replication origins, Nature, 2020, 577(7791):576-581
(2) Li Huang#; Youwang Wang#; Haizhen Long#*; Haoqiang Zhu; Zengqi Wen; Liwei Zhang; Wenhao Zhang; Zhenqian Guo; Longge Wang; Fangyi Tang; Jie Hu; Keyan Bao; Ping Zhu*; Guohong Li*; Zheng Zhou*; Structural insight into H4K20 methylation on H2A.Z-nucleosome by SUV420H1, Molecular Cell, 2023, 83(16): 2884-2895
(3) Yingying Zou#; Jiayao Pei#; Haizhen Long#; Liting Lan; Kejian Dong; Tingting Wang; Ming Li; Zhexuan Zhao; Lirun Zhu; Gangxuan Zhang; Xin Jin; Yang Wang; Zengqi Wen; Min Wei; Yunpeng Feng*; H4S47 O-GlcNAcylation regulates the activation of mammalian replication origins, Nature Structural & Molecular Biology, 2023 Jun;30(6):800-811.
(3) Yan Wang#; Haizhen Long#; Juan yu#; Liping Dong#; Michel Wassef; Baowen Zhuo; Xia Li; Jicheng Zhao; Min Wang; Cuifang Liu; Zengqi Wen; Luyuan Chang; Ping Chen; Qian-fei Wang; Xueqing Xu; Raphael Margueron*; Guohong Li*; Histone variants H2A.Z and H3.3 coordinately regulate PRC2-dependent H3K27me3 deposition and gene expression regulation in mES cells, BMC Biology, 2018, 16(107).
(4) Liuxin Shi; Li Huang; Haizhen Long; Aoqun Song; Zheng zhou*; Structural basis of nucleosomal H4K20 methylation by methyltransferase SET8, FASEB 2022 Jun;36(6)
(5) Ping Chen#, Jicheng Zhao#, Yan Wang#, Min Wang, Haizhen Long, Dan Liang, Li Huang, Zengqi Wen, Wei Li, Xia Li, Hongli Feng, Haiyong Zhao, Ping Zhu, Ming Li, Qian-fei Wang, Guohong Li (2013). "H3.3 actively marks enhancers and primes gene transcription via opening higher-ordered chromatin." Genes Dev. 27, 2109-2124 (2013)
(6) Yongzheng Li#, Boxin Xue#, Mengling Zhang#, Liwei Zhang, Yingping Hou, Yizhi Qin, Haizhen Long, Qian Peter Su, Yao Wang, Xiaodong Guan, Yanyan Jin, Yuan Cao, Guohong Li, Yujie Sun, “Transcription-coupled structural dynamics of topologically associating domains regulate replication origin efficiency”, Genome Biol.2021;22:206